-

-

-
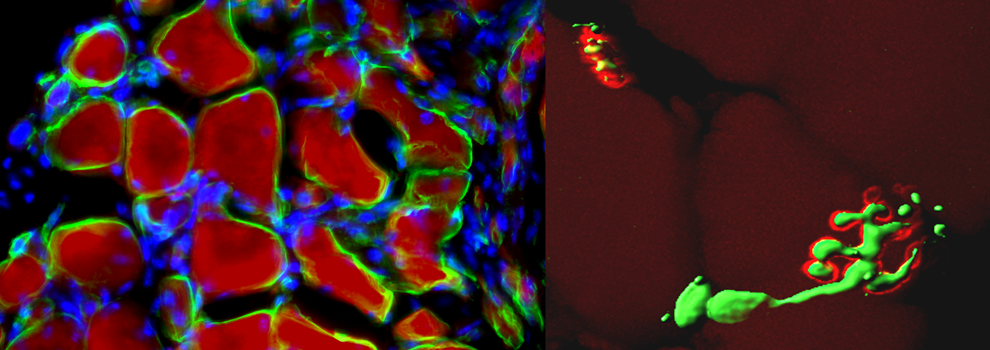
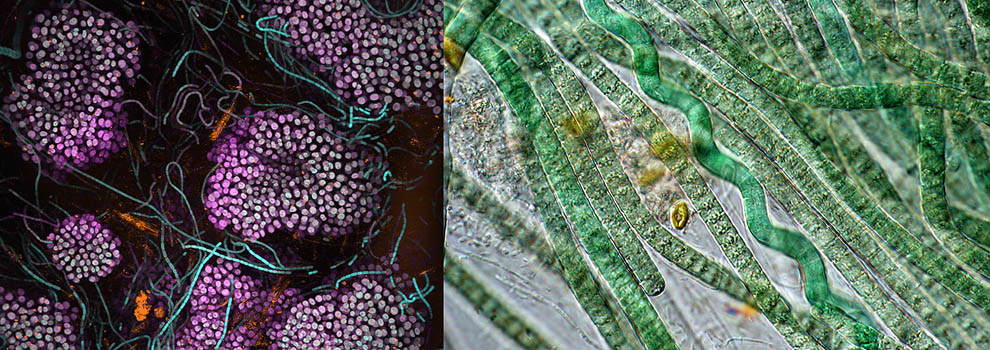
Artificial muscle cross-section:
laminin (green)
Myosin (red)
Dapi (blue)Neuromuscular plaque in
artificial muscle section:
neurofilament (green)
bungarotoxin (red) -

-
 Lab. of Neurochemistry
Lab. of Neurochemistry
Studio dei meccanismi
molecolari delle malattie
neurodegenerative -
 Anemone apennina
Anemone apennina
Monti SimbruiniFoto di Letizia Zanella -
 Studio delle comunità
Studio delle comunità
di batterioplankton nella
Riserva Naturale Regionale
Macchiatonda -
 Astrobiologia e biologia
Astrobiologia e biologia
molecolare di......cianobatteri di
ambienti estremi -
-

Il Dipartimento di Biologia dell’Università degli Studi di Roma “Tor Vergata” orienta la sua missione formativa e di ricerca su tematiche all’avanguardia degli studi sulla vita in tutti i suoi livelli di organizzazione e varietà. Le diverse aree di ricerca concorrono a sviluppare una piattaforma multidisciplinare su temi quali: i meccanismi molecolari delle malattie neurodegenerative, la regolazione dei processi di cancerogenesi; la caratterizzazione di molecole di origine vegetale ed animale; la valutazione delle comunità ecologiche e il monitoraggio ambientale.
Regolamento Dipartimento di Biologia DR 3756 del 06.12.2012
Xle scienze.it, 6/11/2017
Contrariamente a quanto si riteneva fino a qualche tempo fa, la genesi di nuovi neuroni a partire da cellule staminali non si ferma con la fine dell’infanzia, ma continua per tutta l’età adulta, ed è fondamentale per l’apprendimento e la memoria oltre che per la regolazione dell’umore. Uno studio pubblicato su “Cell Stem Cell” da un gruppo di ricercatori dell’Università della North Carolina a Chapel Hill guidati da Juan Song ha ora scoperto che la neurogenesi è controllata da un solo circuito cerebrale. … http://www.lescienze.it/news/2017/11/06/news/centrale_controllo_neurogenesi_cervello_adulto-3742366/
http://www.cell.com/cell-stem-cell/fulltext/S1934-5909(17)30424-1
Nov
AVVISO DI VACANZA
Il Dipartimento di Biologia intende attribuire, mediante affidamento interno, gli insegnamenti vacanti per l’A.A. 2017/2018 come dai seguenti elenchi:
Corso di Laurea Magistrale a Ciclo unico in Farmacia
| Insegnamento |
SSD del corso |
CFU |
semestre |
| Drug Analysis I, Applications II Mod. |
CHIM/08 |
5 |
II anno |
| Cellular and Development Biology II Mod |
BIO/06 |
3 |
I anno |
Il Docente interessato ad assumere l’incarico dovrà segnalare la propria disponibilità entro e non oltre le ore 24:00 del 13 novembre 2017, compilando il modulo della domanda ed inviandolo – tramite e- mail – al Direttore del Dipartimento di Biologia prof.ssa Antonella Canini – canini@uniroma2.it; contestualmente – il docente interessato – informerà il proprio Coordinatore del Corso di Laurea.
N.B.: “Si precisa che il pagamento degli incarichi è subordinato alla disponibilità di bilancio eventuale ed accordata dall’Amministrazione Centrale”.
Nov
wired.it – 2/11/2017, a. pace
Migliaia di informazioni sul funzionamento dei nostri neuroni disponibili online e gratuitamente. Un video per saperne di più
L’idea è dell’Allen institute for Brain science (o scienze del cervello): utilizzare alcune cellule di corteccia cerebrale prelevate durante gli interventi chirurgici, come per esempio la rimozione di un tumore, per incasellare in un unico database più informazioni possibili sulla loro fisiologia e il loro comportamento quando ancora in fase vitale.
Il sistema, online e open access (cioè gratuito), raccoglie le informazioni di centinaia di neuroni provenienti da decine di pazienti differenti, migliaia di dati genetici e molte ricostruzioni in 3D di queste cellule straordinarie ma ancora in parte misteriose, deputate alla conduzione dei segnali elettrici nel cervello. Ed è davvero unico nel suo genere: la maggioranza delle informazioni sulle cellule cerebrali finora ottenute proveniva infatti dallo studio di tessuti morti o dall’indagine su animali da laboratorio, come i topi. … https://www.wired.it/scienza/medicina/2017/11/02/banca-dati-cellule-cervello-vive/
http://celltypes.brain-map.org/
Nov
wired.com, 2/11/2017 – m.musso
Si chiama Earth Microbiome Project, ed è una preziosissima banca dati del microbioma di tutto il nostro pianeta. Il progetto, nato dalla collaborazione di un team di ricercatori provenienti da University of California San Diego, Pacific Northwest National Laboratory, University of Chicago and Argonne National Laboratory, ha raccolto finora oltre 27mila campioni di comunità microbiche provenienti dai più disparati ambienti di tutto il globo, dall’acqua dolce all’acqua salata, fino ad arrivare al suolo della foresta pluviale. E proprio grazie all’analisi del microbioma che vive in ogni singolo campione, come si legge sulle pagine di Nature, i ricercatori sono riusciti a creare la prima banca dati di riferimento dei batteri che colonizzano il pianeta. E siamo solo all’inizio: l’elenco continuerà a crescere man mano che verranno aggiunti nuovi dati. [...] il fondatore del progetto, Rob Knight della San Diego School of Medicine. [...] Per analizzare la diversità batterica tra i vari ambienti, sia geografici che chimici, il team di ricercatori ha sequenziato il gene 16S rRNA, un marcatore genetico specifico per i batteri e i loro parenti, archeobatteri. La sequenza 16S rRNA serve proprio come un codice a barre, ovvero permette di identificare i diversi tipi di batteri. E in questo primo set di dati, i ricercatori hanno identificato ben 300mila sequenze batteriche uniche di 16S rRNA. … http://earthmicrobiome.org/
https://www.nature.com/nature/journal/vaop/ncurrent/full/nature24621.html
https://www.wired.it/scienza/lab/2017/11/02/mappatura-microbioma-pianeta/
PROGETTI:
CERCA NEL SITO:
FACILITIES:
Via della Ricerca Scientifica 1 - 00173 Roma, Tel +39 06 72594391 Fax +39 06 2023500 dipartimentobiologia@pec.uniroma2.it



























Università di Tor Vergata