-

-

-
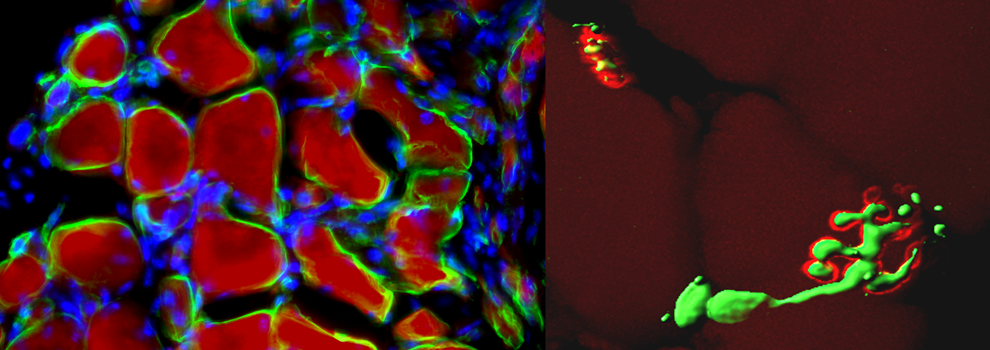
Artificial muscle cross-section:
laminin (green)
Myosin (red)
Dapi (blue)Neuromuscular plaque in
artificial muscle section:
neurofilament (green)
bungarotoxin (red) -

-
 Lab. of Neurochemistry
Lab. of Neurochemistry
Studio dei meccanismi
molecolari delle malattie
neurodegenerative -
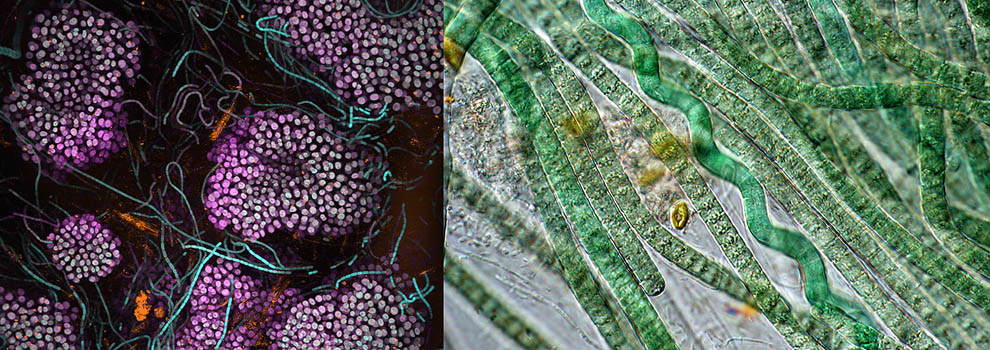
 Anemone apennina
Anemone apennina
Monti SimbruiniFoto di Letizia Zanella -
 Studio delle comunità
Studio delle comunità
di batterioplankton nella
Riserva Naturale Regionale
Macchiatonda -
 Astrobiologia e biologia
Astrobiologia e biologia
molecolare di......cianobatteri di
ambienti estremi -
-

Il Dipartimento di Biologia dell’Università degli Studi di Roma “Tor Vergata” orienta la sua missione formativa e di ricerca su tematiche all’avanguardia degli studi sulla vita in tutti i suoi livelli di organizzazione e varietà. Le diverse aree di ricerca concorrono a sviluppare una piattaforma multidisciplinare su temi quali: i meccanismi molecolari delle malattie neurodegenerative, la regolazione dei processi di cancerogenesi; la caratterizzazione di molecole di origine vegetale ed animale; la valutazione delle comunità ecologiche e il monitoraggio ambientale.
Regolamento Dipartimento di Biologia DR 3756 del 06.12.2012
XOct
la stampa.it – 30/10/2017, n. panciera
Una cura definitiva ancora non esiste, ma la sclerosi multipla vive un’epoca di numerose opzioni terapeutiche e la ricerca di nuove soluzioni è frenetica. La speranza è di arrivare a trattamenti personalizzati perché la sclerosi multipla è una malattia molto eterogenea, sia dal punto di vista clinico che dal punto di vista della risposta ai trattamenti.
Al settimo Congresso Congiunto dei Comitati Europeo e Americano per la Terapia e la Ricerca sulla Sclerosi Multipla (ECTRIMS-ACTRIMS), appena conclusosi a Parigi, si è parlato degli ultimi risultati della ricerca clinica e di base. … http://www.lastampa.it/2017/10/30/scienza/benessere/sclerosi-multipla-cos-la-medicina-si-focalizza-su-trattamenti-pi-concentrati-nel-tempo-Jpr0KWPxVjXC6YQDWd5DzH/pagina.html
Oct
le scienze, 26/10/2017
… Non chiamatelo taglia-incolla. Il sistema CRISPR si è evoluto, e per correggere le mutazioni ora non ha più bisogno di tagliare. Due lavori, pubblicati rispettivamente su “Nature” e su “Science”, annunciano l’arrivo di due nuove varianti di questa piattaforma tecnologica per l’editing genomico. Si tratta di veri e propri correttori automatici di refusi (“base editor”), che prendono il posto delle forbici molecolari della versione classica di CRISPR. [...] L’anno scorso David Liu del Broad Institute aveva già illustrato su “Nature” come convertire una coppia G-C in una coppia T-A. Ma per rimediare a molte mutazioni patologiche serve la transizione inversa e la novità di quest’ultimo lavoro, firmato dallo stesso ricercatore, è che ora è possibile sostituire tutte e quattro le lettere, sia nei batteri che nelle cellule umane. [...] L’altro studio, in uscita su “Science”, è stato eseguito nello stesso istituto, che è nato dalla collaborazione tra Massachusetts Institute of Technology e Harvard University, ed è firmato da uno dei pionieri di CRISPR: Feng Zhang. La nuova variante ha ricevuto il nome REPAIR (RNA Editing for Programmable A to I Replacement) … http://www.lescienze.it/news/2017/10/26/news/crispr_dna_rna-3729557/
https://www.nature.com/nature/journal/vaap/ncurrent/full/nature24644.html
http://science.sciencemag.org/content/early/2017/10/24/science.aaq0180
Scienza in rete.it – 30/10/2017 – https://www.scienzainrete.it/articolo/nuova-crispr-base-base/laura-mosca/2017-10-30
le scienze.it, 26/10/2017
Per la prima volta un gruppo internazionale di ricercatori è riuscito a fornire una misura generale dell’efficacia degli sforzi per la conservazione della biodiversità. Gli scienziati in particolare hanno mostrato che, grazie agli investimenti in questo campo, nei 109 paesi che nel 1992 hanno firmato la Convenzione sulla biodiversità (nota anche come Convenzione di Rio) fra il 1996 e il 2008 il tasso di perdita delle specie è calato del 29 per cento. Inoltre lo studio – pubblicato su “Nature” – fornisce uno strumento che permette alle nazioni di calibrare i propri investimenti nella conservazione in base loro specifiche esigenze.[...]
Analizzando i compromessi tra conservazione e sviluppo economico, i ricercatori sono stati in grado di creare un algoritmo che suddivide la responsabilità del declino delle specie globali tra i singoli paesi e stima come adeguare i bilanci finanziari per la conservazione per bilanciare i tassi di crescita economica umana attuali o futuri. [...] Sulla base di questo algoritmo, i ricercatori hanno infine stimato che con un’efficiente distribuzione delle risorse per la conservazione, basterebbe meno dello 0,01 per cento del prodotto interno lordo globale per fermare la perdita di biodiversità. https://www.nature.com/nature/journal/vaop/ncurrent/full/nature24295.html
http://www.lescienze.it/news/2017/10/26/news/riduzione_perdita_biodiversita_investimenti_sviluppo-3728903/
Oct
la repubblica.it, 25/10/2017 – s. iannacone
Si chiama Repair, acronimo di Rna Editing for Programmable A to I Replacement. Ed è una nuova tecnica di ingegneria genetica, i cui dettagli sono stati appena pubblicati sulle pagine di Science, che consentirà di agire direttamente sull’Rna, il “gemello diverso” del Dna, prodotto nel processo di trascrizione del codice genetico e protagonista cruciale della sintesi delle proteine. La tecnica è stata messa a punto da un’équipe di scienziati del Broad Institute e del Massachusetts Institute of Techonology (Mit) e fa parte della “famiglia” Crispr, [...] la ricerca appena pubblicata mostra che, fatte le opportune modifiche, è possibile utilizzare Crispr anche per agire su singole “lettere” dell’Rna. Il che, dicono gli autori del lavoro, spiana la strada a nuovi fronti di ricerca e applicazioni cliniche finora del tutto inesplorati. … http://science.sciencemag.org/content/early/2017/10/24/science.aaq0180
http://www.repubblica.it/scienze/2017/10/25/news/genetica_la_famiglia_crispr_si_allarga_da_oggi_e_possibile_modificare_anche_l_rna-179325361/
PROGETTI:
CERCA NEL SITO:
FACILITIES:
Via della Ricerca Scientifica 1 - 00173 Roma, Tel +39 06 72594391 Fax +39 06 2023500 dipartimentobiologia@pec.uniroma2.it




























Università di Tor Vergata