-

-

-
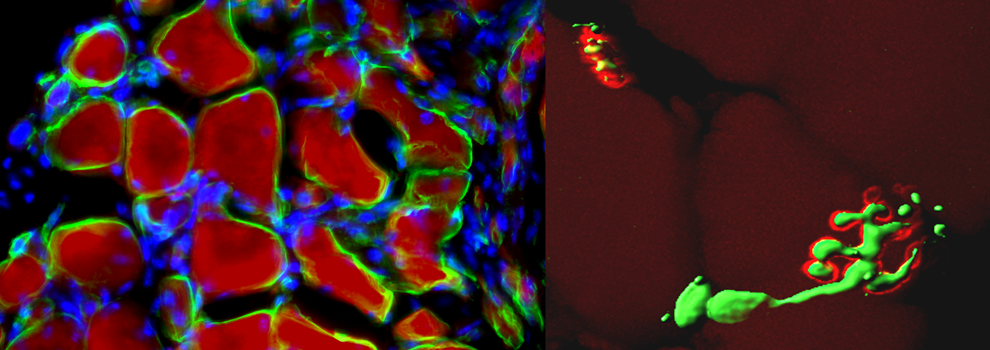
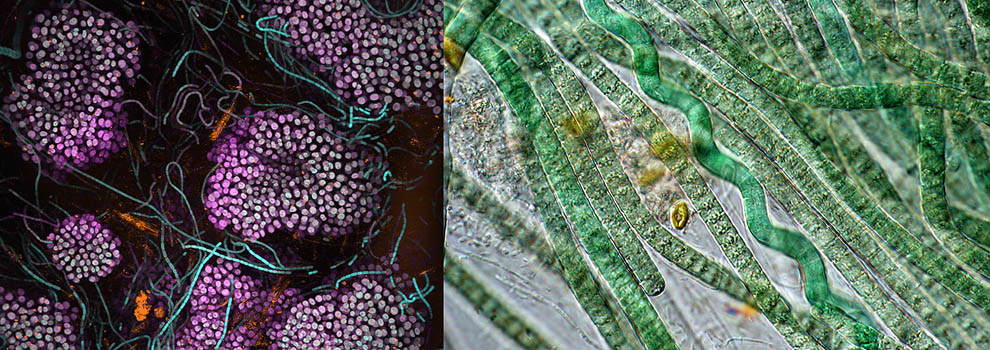
Artificial muscle cross-section:
laminin (green)
Myosin (red)
Dapi (blue)Neuromuscular plaque in
artificial muscle section:
neurofilament (green)
bungarotoxin (red) -

-
 Lab. of Neurochemistry
Lab. of Neurochemistry
Studio dei meccanismi
molecolari delle malattie
neurodegenerative -
 Anemone apennina
Anemone apennina
Monti SimbruiniFoto di Letizia Zanella -
 Studio delle comunità
Studio delle comunità
di batterioplankton nella
Riserva Naturale Regionale
Macchiatonda -
 Astrobiologia e biologia
Astrobiologia e biologia
molecolare di......cianobatteri di
ambienti estremi -
-

Il Dipartimento di Biologia dell’Università degli Studi di Roma “Tor Vergata” orienta la sua missione formativa e di ricerca su tematiche all’avanguardia degli studi sulla vita in tutti i suoi livelli di organizzazione e varietà. Le diverse aree di ricerca concorrono a sviluppare una piattaforma multidisciplinare su temi quali: i meccanismi molecolari delle malattie neurodegenerative, la regolazione dei processi di cancerogenesi; la caratterizzazione di molecole di origine vegetale ed animale; la valutazione delle comunità ecologiche e il monitoraggio ambientale.
Regolamento Dipartimento di Biologia DR 3756 del 06.12.2012
XOct
wired.it, 19/10/2017, m. magistroni
La Food and Drug Administration (Fda) ha approvato l’utilizzo di una nuova terapia anti-cancro, la seconda negli Stati Uniti. Yescarta (axicabtagene ciloleucel) è un trattamento che utilizza i linfociti T del paziente modificati in laboratorio per attaccare le cellule del tumore. Il nullaosta riguarda la cura di pazienti adulti colpiti da linfomi a cellule B non-Hodgkin di grandi dimensioni che non rispondono alle terapia standard o che hanno subito delle ricadute.
Quella messa a punto da Kite Pharma è una strategia di immunoterapia, una metodologia che trasforma le cellule del sistema immunitario del paziente in killer del tumore. …
http://www.lescienze.it/news/2017/10/19/news/fiori_alone_blu_interferenza_api_impollinatori-3715931/
https://www.fda.gov/NewsEvents/Newsroom/PressAnnouncements/ucm581216.htm
http://www.lastampa.it/2017/10/19/scienza/benessere/nuova-terapia-genica-dagli-usa-trasforma-le-cellule-del-paziente-in-killer-del-cancro-mrj05RpxsdypVzNPoEumLJ/pagina.html – f. di todaro
le scienze.it – 19/10/2017
Per attirare gli impollinatori, molti fiori presentano microscopiche creste sulla superficie dei loro petali che, a prescindere dal colore di base del fiore, diffondono un “alone blu” a cui gli insetti sono particolarmente sensibili. A scoprilo è stato un gruppo di ricercatori dell’Università di Cambridge che firmano un articolo su “Nature”. [...] studi sulla sensibilità spettrale dei fotorecettori delle api hanno anche scoperto che questi insetti sono relativamente insensibili alla maggior parte dei colori, con l’eccezione del blu e dell’ultravioletto.
Osservando i petali di fiori di 12 specie evolutivamente distanti, Edwige Moyroud e colleghi hanno rilevato la presenza di increspature e scanalature nanometriche in grado di provocare un fenomeno di interferenza analogo a quello che provoca l’iridescenza delle ali delle farfalle, che produce attorno alle parti del petalo un alone di luce nella banda fra il blu e l’ultravioletto. … https://www.nature.com/nature/journal/vaop/ncurrent/full/nature24285.html
http://www.lescienze.it/news/2017/10/19/news/fiori_alone_blu_interferenza_api_impollinatori-3715931/
la stampa.it, 18/10/2019 tuttoscienze, n. panciera
Il Nobel Hoffmann: da loro abbiamo ereditato i meccanismi immunitari più sofisticati. Adesso li studiano gli americani Allison e Schreiber per battere i tumori.
…il Premio Nobel per la medicina al biologo lussemburghese Jules Hoffmann. «Quando abbiamo iniziato a studiarli, sapevamo che questi animali possiedono una relativa resistenza, ma non ne conoscevamo i meccanismi», racconta. Ha quindi scoperto che si tratta di una forma di immunità innata. Ma ciò che nessuno sospettava è che questa caratteristica è presente in tutte le specie di insetti e nei mammiferi: è un sistema che, dai moscerini della frutta studiati da Hoffmann fino all’uomo, si è conservato nei tempi lunghi dell’evoluzione. Hoffmann, direttore di ricerca emerito del Centre National de la Recherche Scientifique all’Istituto di Biologia molecolare e cellulare a Strasburgo, è stato ospite a Milano in occasione dell’annuncio dell’edizione 2017 del Premio Balzan: circa 660 mila euro, metà dei quali da destinarsi a progetti condotti da giovani ricercatori. Al biologo, già insignito del Balzan nel 2007, è spettato il compito di presentare i vincitori di quest’anno per le scienze mediche: sono James Allison della University of Texas e Robert Schreiber della Washington University, le cui ricerche di frontiera riguardano proprio il sistema immunitario e il ruolo nel contrastare il cancro. [...] l’identificazione di una serie di antigeni associati al cancro è frutto del lavoro di Schreiber e Allison, i quali riceveranno il Premio Balzan nel corso della cerimonia a Berna il 17 novembre. Allison ha studiato i meccanismi in grado di bloccare la risposta immunitaria, i «checkpoint» immunitari. A lui si deve l’idea di inibirli per dare slancio ai linfociti T attraverso l’uso di anticorpi monoclonali. Così l’immunologo ha aumentato, per la prima volta, la sopravvivenza in pazienti affetti da melanoma metastatico. A svelare i meccanismi molecolari alla base della lotta tra cancro e sistema immunitario (l’«immuno-editing») è stato, invece, Schreiber, che ne ha svelato le fasi di eliminazione, equilibrio e fuga immunitaria, nelle quali il tumore, rispettivamente, viene eradicato, rimane in equilibrio o si sottrae al sistema immunitario. … http://www.lastampa.it/2017/10/18/scienza/tuttoscienze/la-forza-degli-insetti-sia-con-noi-kVkDT1CRD0cAR6zdv0R2oO/pagina.html
Oct
oggiscienza.it, 17/10/2017, m. turconi
… il tema del neuroenhancement, ossia del potenziamento cognitivo, è un filone di indagine che abbraccia diversi campi delle neuroscienze, spaziando dalla farmacologia alle neuroscienze cognitive, e che interessa decine di gruppi di ricerca in tutto il mondo. Questo perché, oltre a migliorare le funzioni cognitive di individui sani, identificare strategie efficaci di potenziamento cognitivo aiuterebbe a contrastare gli effetti devastanti di patologie neurodegenerative, sempre più diffuse a causa dell’aumento dell’aspettativa di vita media e dall’impatto socioeconomico devastante.
Ed è proprio di questi giorni la notizia di uno studio, realizzato da un gruppo della Boston University, che spiega come un’innovativa tecnica di stimolazione neurale (la high-definition transcranial alternating current stimulation, o HD-tACS) possa contribuire al rafforzamento delle funzioni esecutive, abilità cognitive cruciali per comportamenti complessi come l’autocontrollo, la coordinazione dei movimenti, la memoria. … La HD-tACS, a differenza di quanto il nome potrebbe far pensare, è una metodica non invasiva con la quale si altera, in maniera del tutto indolore, l’attività dei neuroni tramite l’applicazione di una corrente elettrica alternata. Nello specifico, il team di ricerca guidato dal Professor Robert Reinhart ha agito su due aree della corteccia cerebrale adibite al controllo delle funzioni esecutive, quella mediale frontale e quella laterale prefrontale: l’obiettivo era quello di sincronizzare le oscillazioni (ossia le attivazioni di gruppi di neuroni) tra queste due aree, migliorando così la loro connessione e, di conseguenza, i processi cognitivi da esse governate. [...] Lo studio, pubblicato sui Proceedings of the National Academy of Sciences (PNAS), ha dimostrato come l’aumento di sincronizzazione possa migliorare le performance di compiti di laboratorio; analogamente, Reinhart e colleghi hanno dimostrato che la desincronizzazione dell’attività si traduce in risultati peggiori. …
http://www.pnas.org/content/early/2017/10/03/1710257114
PROGETTI:
CERCA NEL SITO:
FACILITIES:
Via della Ricerca Scientifica 1 - 00173 Roma, Tel +39 06 72594391 Fax +39 06 2023500 dipartimentobiologia@pec.uniroma2.it





























Università di Tor Vergata