-

-

-
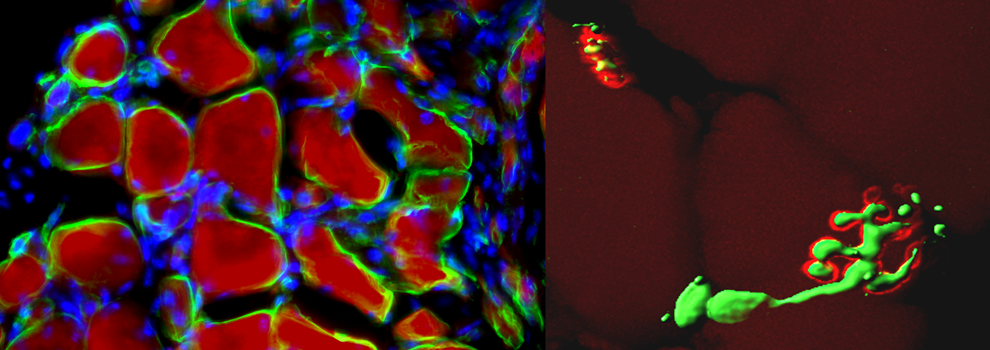
Artificial muscle cross-section:
laminin (green)
Myosin (red)
Dapi (blue)Neuromuscular plaque in
artificial muscle section:
neurofilament (green)
bungarotoxin (red) -

-
 Lab. of Neurochemistry
Lab. of Neurochemistry
Studio dei meccanismi
molecolari delle malattie
neurodegenerative -
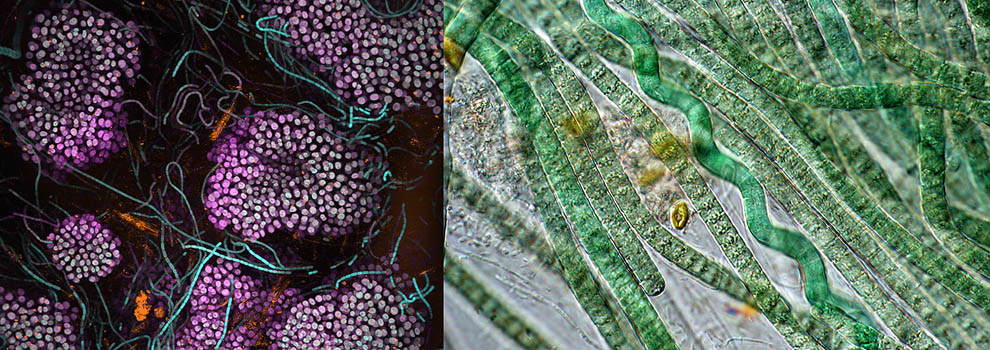
 Anemone apennina
Anemone apennina
Monti SimbruiniFoto di Letizia Zanella -
 Studio delle comunità
Studio delle comunità
di batterioplankton nella
Riserva Naturale Regionale
Macchiatonda -
 Astrobiologia e biologia
Astrobiologia e biologia
molecolare di......cianobatteri di
ambienti estremi -
-

Il Dipartimento di Biologia dell’Università degli Studi di Roma “Tor Vergata” orienta la sua missione formativa e di ricerca su tematiche all’avanguardia degli studi sulla vita in tutti i suoi livelli di organizzazione e varietà. Le diverse aree di ricerca concorrono a sviluppare una piattaforma multidisciplinare su temi quali: i meccanismi molecolari delle malattie neurodegenerative, la regolazione dei processi di cancerogenesi; la caratterizzazione di molecole di origine vegetale ed animale; la valutazione delle comunità ecologiche e il monitoraggio ambientale.
Regolamento Dipartimento di Biologia DR 3756 del 06.12.2012
Xla scienze.it, 5/12/2017
Ma come fa la mente umana ad avere un controllo preciso e flessibile degli intervalli di tempo in cui svolgere un compito? Ora un gruppo di ricercatori del Massachusetts Institute of Technology propone sulle pagine di “Nature Neuroscience” un nuovo modello di come il cervello sovraintende alle attività che richiedono una precisa tempistica.
http://www.lescienze.it/news/2017/12/05/news/neuroni_tengono_tempo-3779826/
https://www.nature.com/articles/s41593-017-0028-6
Dec
oggiscienza – 6/12/2017, e. degano
Sono più di 350 000 le specie vegetali che usano semi per riprodursi, oltre il 90% della flora sul pianeta…
Thor Hanson, biologo della conservazione e scrittore, nel suo «Semi. Viaggio all’origine del mondo vegetale» da poco pubblicato nell’edizione italiana (il Saggiatore…) https://oggiscienza.it/2017/12/06/semi-thor-hanson-saggiatore/
Dec
le scienze – 4/12/2017
Alcuni licheni modificano la loro strategia riproduttiva in base alle condizioni meteorologiche. Lo studio che lo ha provato – condotto da un gruppo di ricercatori dell’Università Rey Juan Carlos a Madrid e pubblicata sugli “Annals of Botany” – amplia così l’ambito degli organismi per i quali vale la cosiddetta teoria delle strategie di vita (life-history theory). … http://www.lescienze.it/news/2017/12/04/news/strategia_variabile_riproduzione_licheni-3778664/
https://www.botany.one/2017/08/reproductive-strategy-lichenized-fungus-shifts-along-climatic-gradient/
scienzainrete.it, 1/12/2017 – l. mosca
… il meccanismo di trasporto attraverso le cellule eucariote è stato oggetto di uno studio di recente pubblicazione apparso sulla rivista scientifica mBio ad opera dei ricercatori della Monash University a Melbourne, in Australia coordinati dall’esperto di fagi Jeremy Barr.
Nel loro lavoro gli studiosi hanno osservato che le cellule epiteliali assorbono e trasportano i fagi attraverso la cellula mediante transcitosi, cioè il processo cellulare tramite il quale la cellula trasporta le macromolecole da un lato all’altro attraverso il citoplasma; in questo modo i fagi attivi vengono liberati sulla superficie opposta della cellula. Mediante studi in vitro i ricercatori hanno dimostrato che la transcitosi di diversi batteriofagi avviene in maniera rapida e direzionale attraverso strati di cellule confluenti provenienti da intestino, polmone, fegato, rene e cervello. La transcitosi dei batteriofagi attraverso gli strati cellulari ha mostrato… http://www.scienzainrete.it/articolo/fagoma-fa-differenza/laura-mosca/2017-12-01
http://mbio.asm.org/content/8/6/e01874-17.abstract?sid=ea9a19b0-3923-4163-bb52-e5c6b9e4de90
PROGETTI:
CERCA NEL SITO:
FACILITIES:
Via della Ricerca Scientifica 1 - 00173 Roma, Tel +39 06 72594391 Fax +39 06 2023500 dipartimentobiologia@pec.uniroma2.it





























Università di Tor Vergata